효소 유형 사용해 절단할 유전체 표적 부위 뿐 아니라 편집할 DNA도 지정

|
노벨상을 수상한 유전자 편집 기술인 CRISPR보다 유전 공학에 훨씬 더 유용할 수 있는 유전자 편집기술이 국제 학술지 네이처에 6월 26일 두편의 논문으로 공개됐다.
미국 및 일본 연구진들이 DNA를 재조합, 재배열할 수 있는 ‘브릿지(bridge) 재조합’ 메커니즘을 발견했다는 연구 결과다.
기존에는 크리스퍼(CRISPR) 유전자 편집기술이 있었다. CRISPR은 효소를 사용해 원하는 위치에서 DNA를 절단한 후 세포의 복구 메커니즘이 작동해 DNA를 변경할 수 있어 단일 유전자에 여러 돌연변이가 있는 환자들을 치료하거나 면역세포가 여러 가지 방식으로 암을 공격하도록 하는 등 의학적 용도로 적용되고 있다.
다만, 엉뚱한 부분을 자르거나 세포가 복구를 수행하는 동안 의도하지 않은 부수적인 유전적 손상이 있을 수 있었다.
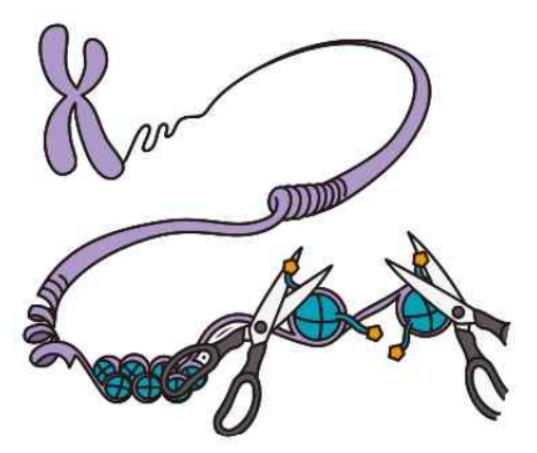
반면 네이처에 발표된 새로운 ' 브릿지 유전자 편집 시스템' 은 RNA 조각과 재조합효소단백질이라고 불리는 효소 유형을 사용해 절단할 유전체 표적 부위 뿐 아니라 편집할 DNA도 지정할 수 있다.
연구진에 따르면 이 체계는 거의 어떤 길이든지 DNA 순서를 삽입하거나, 제거하거나 역위(invert)하기 위해 이용될 수 있다.
연구진은 이 시스템을 사용해 거의 5,000개의 염기 길이를 가진 DNA 조각을 대장균 박테리아 유전체에 정밀하게 삽입하고 대장균 유전체에서 또 다른 DNA 조각을 절제하고 역위시켰다.
인간 세포에 작동한다면 ‘혁명적’...박테리아 외 다른 세포에서 작동하며 ‘게임 체인저’
새로운 유전자 편집 도구를 찾기 위해 연구진들은 박테리아의 이동성 DNA 요소가 한 위치에서 다른 위치로 이동할 수 있도록 하는 다양한 종류 효소를 선별했다. 그리고 박테리아 유전체에서 발견된 점핑 유전자(jumping gene)인 IS110은 다른 위치로 이동할 때 DNA에서 자신을 잘라낼 때 브릿지 RNA를 만드는 것을 발견했다. IS110 계열 효소는 복잡하고 특이한 RNA 기반 표적화 시스템을 사용한다고 연구팀은 밝혔다.
RNA 한쪽 끝은 유전체에 삽입될 DNA 조각에 결합하고 다른 쪽 끝은 이동할 유전체 부위 DNA 조각에 결합한다. RNA가 두 DNA 세그먼트를 연결하기 때문에 연구팀은 이 분자를 '브릿지 RNA'라고 명명했다.
이 두 편의 논문과 함께 발표된 네이처 논평에 따르면, 이 기술은 박테리아에서만 테스트 됐으나 이것이 인간 세포에서 작동한다면 그것은 ‘혁명적’일 수 있다고 평가된다.
호주 시드니 대학 구조 생물학자이자 Nature Communications 저자인 Sandro Fernandes Ataide도 “이것이 다른 세포에서 작동한다면, 게임 체인저가 될 것”이라며 “유전자 편집의 새로운 분야를 열고 있다”고 평가했다.
이에 대해 한국바이오협회는 "'브릿지 RNA‘ 또는 'seekRNA'라고 불리는 RNA 분자에 의해 유도되는 이 시스템은 박테리아와 시험관 반응에서 유전자를 편집하는 것으로 나타났지만 인간 세포에서 작동하도록 적응할 수 있는지 여부는 여전히 불분명하다"며 " 만약 그것이 가능하다면, 그것은 작은 크기와 DNA를 파괴하지 않고 수천 개의 염기(CRISPR-Cas9 유전체 편집 시스템에서 실용가능한 것보다 훨씬 더 큼)의 유전적 변화를 일으킬 수 있는 능력으로 인해 혁명적일 수 있다"고 전했다.
-
01 [신년사] "'국민 안전·산업에 힘이 되는 규... -
02 제니텍 홍성창 회장,'2026년 한국을 이끌 혁... -
03 "제약바이오산업과, 현장 목소리 신속하고 ... -
04 [신년사] 제약바이오헬스산업 경쟁력 강화 ... -
05 [신년사]"약가 개편 등 위기… 290개 회원사 ... -
06 [신년사] "K-바이오, 추격자 넘어 혁신 주체... -
07 [신년사]革故鼎新(혁고정신), 2026년 제약 ... -
08 ★대상 부산오거리약국 황은경약사 -
09 [신년사] "유통의 가치가 재조명되는 전환의... -
10 [신년사] "행동·실천으로 회원과 함께 여는 ...











